4个回答
|
我不太清楚你输的是什么序列,我大概说一下可能的问题。1.他要求同时输入两种序列,一种是你的PCR产物的测序结果,一般是seq格式或者是多个seq格式的压缩文件,还有一种是你PCR产物对应的基因组序列,即未亚硫酸氢盐处理的序列,作为模版与你的测序序列相比较。2.他在分析时有一系列的指标,比如mismatch不能超过多少,亚硫酸氢盐转化效率不能低于多少等等,如果你的测序结果经过分析不符合这些标准也不能产生图。不知道这些能不能帮到你,或者你可以传一下你输入序列后产生的截图,这样更方便看是什么问题。 |
|
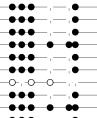
BIQ我没用过,我一般用QUMA出图,我的图中一般也会有黑白圈中间有时也会出X,代表碱基突变,因为我不是用的高保真酶扩增的,你可以自己手工比较你的测序结果和原始的gDNA亚硫酸氢盐处理后的序列,比较CG位点是否有突变 |
|
你上传的序列全部被excluded了啊,不符合它的要求,你看一下你的转化效率,或者是否错配太多?你把下面那部分的图传一下,那部分图上面标题写了亚硫酸氢盐效率,等一系列标题,你看是哪个不符合要求 |
你的回答
| 提问时间: | 2024-04-13 |
| 浏览量: | 4048 |
| 最近回答: | 2024-04-13 |
相关问题
{title}
投票 {vote}