|
2024-04-13 14:18:48
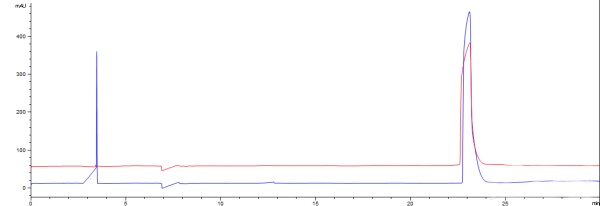
CE-SDS和SDS-PAGE分析蛋白纯度的差异CE-SDS和SDS-PAGE都是用于检测蛋白纯度的电泳方法,药典也都有记载,算很常规的操作。 但是最近我在做一个蛋白(无糖基化)纯度分析时就发现了这么个问题。还原(红色)和非还原(蓝色)CE-SDS结果见下图,我这里已经把蛋白提高到20 g/L,最终上样的浓度是5 g/L。积分下来纯度都是100%,出峰时间几乎一样。也就是说,还原和非还原分子量一样,而且没看到杂带。
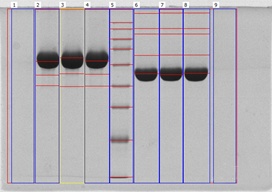
下面这个是SDS-PAGE结果,从左到右依次为3个还原操作,Marker,3个非还原操作。点样浓度10ug/孔。胶浓度10%。
两个一对比就发现问题了,第一CE-SDS的纯度是100%,是怎么改积分参数都没有其他峰积出来,而SDS-PAGE结果扫描软件就能扫到杂带,非还原有部分聚体,还原有一些低分子量的。第二是SDS-PAGE怎么非还原的分子量要比还原的要小?而CE-SDS是两个出峰时间是一样的。 针对第一个问题,这是CE的灵敏度没有SDS-PAGE高吗?还是SDS-PAGE的杂带是前处理反应产生的?因为这个方法也经常跑其他单抗项目的CE-SDS,NGHC和HC这种分子量只差几道尔顿的都能分离,SDS-PAGE得到的杂带比主条带都要差10道尔顿以上了,理论上对CE应该是能分离出来的。 针对第二个问题,我这边前处理条件都是一样的,仅仅是buffer不同,CE-SDS用的是贝克曼里面的buffer,SDS-PAGE的loading buffer是根据药典配的。这些参数和buffer平时跑单抗没什么问题的,分子量都是对的,杂带也基本和CE-SDS对的上。而这次SDS-PAGE只有还原条带分子量和理论是一样的,非还原条带少了将近10道尔顿。 请教各位大神,这怎么解释,我该信哪个的结果?
|
2个回答
|
第一个问题:差异是来源于两种方法的上样量。PAGE(100ug)的绝对量是CE(10~20ng)的接近5000~10000倍,这本身就已经把杂质的量绝对放大了,但是对于单体来说只是颜色加深了,峰宽和峰面积不会有太大的变化,这样相当于是人为的提高了杂质的比例。这个结果其实是有偏差的。所以在现在的方法体系里面page方法是半定量方法。而CE是定量方法。 第二个问题:我觉得来源于两种方法的胶的差别,CE是线性胶,PAGE是非线性胶。非线性胶的分子筛效应比线性胶要高。当蛋白的二硫键比较少的时候,对于CE方法来说,还原非还原的差别没有那么明显,质荷比接近,体积因素没有影响太多。而对于page,由于分子筛效应较大,体积因素就有较大影响了。 |
你的回答
| 提问时间: | 2024-04-13 |
| 浏览量: | 4057 |
| 最近回答: | 2024-04-13 |