|
2024-04-08 15:04:16
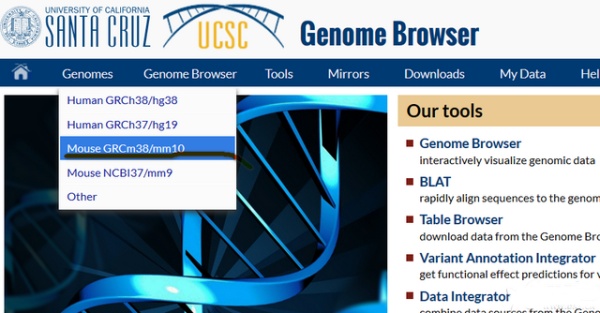
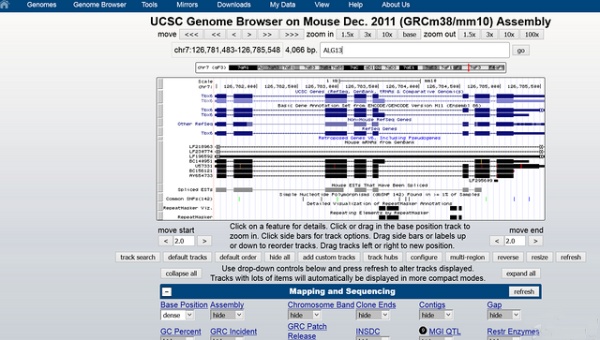
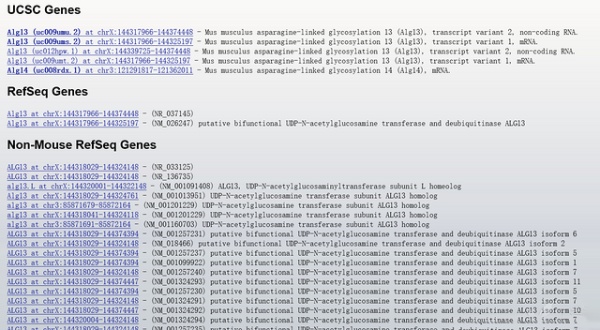
如何在UCSC查找基因的DNA全序列?想查一个基因的DNA全序列(包括启动子、3’-UTR等等),经推荐UCSC,进去一查,傻眼了,那么多信息,什么是我需要的啊?!!都说UCSC很强大,可是作为一般人,貌似很多功能都用不到吧,太多信息充斥在页面里面,一点都没有NCBI好,需要什么功能,都列表列的清清楚楚,界面也很清爽,而UCSC,怎么辣么多选项/琐碎信息??求助大侠看看这下面的界面怎么使用,我看的头昏眼花。。 比如:我要查小鼠 这个基因 ALG13 的全DNA序列(包括启动子、3’-UTR等等) 第一步: 第二步: (这个界面我只知道输入基因名称,中间的好几个直线图是代表什么意思? 还有,下面的几十个选项有什么用途?) 第三步:(冒出这么多信息,看的眼花) 所以,求助各位大侠,我只是一个小小的愿望(查询某个基因的DNA全长),怎样灵活地使用UCSC ?谢谢! |
7个回答
|
可以在定位框中输入基因名称,也可以直接输入核苷酸的坐标值,例如SNP的rs号。碱基位置中间几个直线图是UCSC的特色:可视化缩放条(包括染色体表意图和好多track的示意图)。下面几十个选项是用户自定义显示项目,用户可以选择缩放条的缩放倍数,反向显示序列,移动当前显示的序列物理位置等等,修改显示后点refresh按钮刷新显示。现在的UCSC网站是改版之后的,以前的UCSC界面还算清爽。 |
|
这个其实和NCBI里的一样,你的最后一张图上Refseq就是呀,NM_026247就是在转剧本,点进去应该能找到 |
|
都是一个基因的不同转录本,mRNA基本很大部分都是相同的,两个转录本,gene列表里1和3都是非编码RNA,一般研究不会做到不同转录本那么细致,选其中一个转录本就就可以了 |
|
我一般习惯用NCBI,最近学了用UCSC看组蛋白的甲基化情况,然后就是看基因的启动子,其余的一般都用NCBI看 |
|
UCSC除了可以查启动子外显子内含子,还可以①定位源序列在基因组中的位置,②查找源序列的上、下游基因,③查看基因组注释信息(如CpG岛,EST等),④BLAT和in Silico PCR搜索序列等等。既然叫基因组浏览器,主要是以网页分析工具、浏览定位序列 为主要功能。。NCBI和Ensembl 的使用广泛程度还是比UCSC高得多,用不习惯UCSC就试试NCBI 或Ensembl ,都差不多。 |
你的回答
| 提问时间: | 2024-04-08 |
| 浏览量: | 4123 |
| 最近回答: | 2024-04-08 |