郑州大学开发新型基因编辑工具 提高基因编辑效率3至4倍
2018年7月10日,郑州大学宋永平课题组等人在Cell Discovery上在线发表了题为“Genetic editing and interrogation with Cpf1 and caged truncated pre-tRNA-like crRNA in mammalian cells”的研究论文。研究人员报道了一种Rnase抗性的pre-tRNA-like crRNA (catRNA),它可以利用Lachnospiraceae细菌Cpf1(LbCpf1)进行精确有效的基因编辑。 与常规crRNA诱导的相比,catRNA的特异性基因敲除和敲入分别增加了3.2倍和4.3倍。该研究揭示了catRNA的潜力和CRISPR / Cpf1系统的通用应用,为哺乳动物细胞中的选择性基因扰动建立了一种简单的方法。

Cpf1是一种新型的II类CRISPR系统组件,具有与Cas9不同的特征,是一种单一RNA引导的核酸内切酶,可识别富含胸苷的原型间隔区相邻基序(PAM)并在PAM位点远端产生交错切。 这种V型CRISPR / Cpf1系统在哺乳动物细胞、以及动物,植物和细菌中表现出强大的基因组编辑活性。 有趣的是,Cpf1是一种双核酸酶,不仅可以切割靶DNA,还可以处理自身的CRISPR RNA(crRNA)。 此外,Cpf1对crRNA的成熟不需要反式激活crRNA(tracrRNA)的帮助。 由于具有这些优点,Cpf1系统最近被用于哺乳动物和植物细胞中的多重基因编辑,其中Cpf1使用由成熟直接重复12,13间隔的单个crRNA阵列同时编辑多达四个基因。
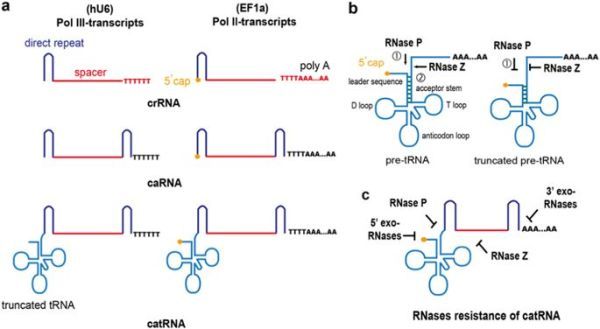
设计Rnase抗性的pre-tRNA-like crRNA (catRNA)
与Cas9的高脱靶潜力相比,全基因组深度测序揭示了Cpf1非常精确的基因破坏,使得Cpf1成为Cas9的理想基因修饰替代物。尽管Cpf1在哺乳动物细胞中介导有效的基因编辑,但其整体活性不如Cas9那样强大。研究人员推测crRNA部分地降低了其降低的基因破坏效率,因为crRNA中存在的茎环结构较少,比sgRNA中的各种茎环提供的RNA酶抗性更少。tRNA是最稳定的RNA分子,因为它们具有多个发夹(或茎环)和3'ploy尾结构,其赋予对各种RNase分子的抗性。pre-tRNA偶尔在其5'末端用甲基鸟苷封端。最近在酿酒酵母中进行的一项研究发现,甲基鸟嘌呤帽结构可保护前tRNA免受RNase的降解,并且帽结构可能在成熟过程中起到保护pre-tRNA的屏障的作用。
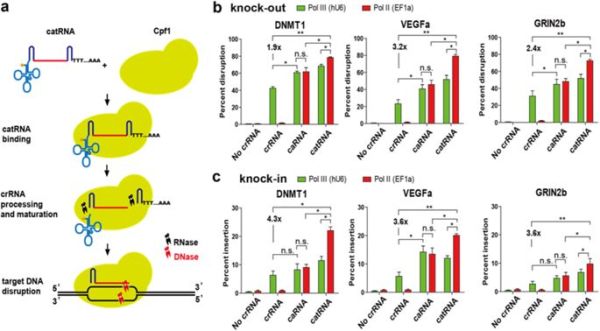
catRNA增强哺乳动物细胞的敲除和敲入
在此,研究人员报道了一种Rnase抗性的pre-tRNA-like crRNA (catRNA),它可以利用Lachnospiraceae细菌Cpf1(LbCpf1)进行精确有效的基因编辑,并能够将缺乏DNA核酸内切酶活性的LbCpf1(dCpf1)重编程为转录调节因子。 与常规crRNA诱导的相比,catRNA的特异性基因敲除和敲入分别增加了3.2倍和4.3倍。 当使用电穿孔时,观察到更高的基因破坏增加(高达37倍)。 研究人员在此报道catRNA能够通过dCpf1激活剂进行有效的基因激活。该研究揭示了catRNA的潜力和CRISPR / Cpf1系统的通用应用,为哺乳动物细胞中的选择性基因扰动建立了一种简单的方法。
来源:iNature
版权及免责声明:本网站所有文章除标明原创外,均来自网络。登载本文的目的为传播行业信息,内容仅供参考,如有侵权请联系答魔删除。文章版权归原作者及原出处所有。本网拥有对此声明的最终解释权。


{replyUser1} 回复 {replyUser2}:{content}