中外学者报道六种分析表观基因组、转录组和蛋白质组的新技术
现阶段,科学家们已经开始以单细胞分辨率组合多层信息。这些“多组学”技术可以更仔细地观察细胞之间的可变性,更清楚地识别特定细胞及其功能。分析基因组DNA揭示了单细胞基因组,甲基化组织或染色质,而分析RNA和蛋白质则能分别产生转录组和蛋白质组数据。
表观基因组和转录组
分析细胞表观基因组和转录组的技术可以揭示甲基化和染色质可接近性(chromatin accessibility)对基因表达的调控作用。
“在肿瘤发生等复杂的生物过程中,异质性同时存在于基因组,表观基因组和转录组中,单独分析它们可能不起作用,”北京大学分子生物学家汤富酬教授说。基因相同的肿瘤细胞可能具有不同的DNA甲基化或基因表达模式,可能需要多组学技术才能明确地将它们分类为亚群。
scM&T-seq(simultaneous single-cell methylome and transcriptome sequencing)能同时完成单细胞甲基化组和转录组测序分析,这种方法基于G&T-seq,采用相同的方法从单个细胞中分离DNA和RNA,并扩增和测序RNA。对基因组DNA进行亚硫酸氢盐处理,将未甲基化的胞嘧啶转化为尿嘧啶,然后扩增并测序以测定甲基化组。
研发技术人员表示,“这一新的实验方案让你可以并行分析同一单细胞中的DNA甲基化和RNA。我们的方法提供了有关单细胞DNA甲基化异质性与特定基因表达差异之间关系的首个直观视图。”(Nature Methods:突破性单细胞表观基因组与转录组分析新技术)
scNMT-seq(single-cell nucleosome, methylation, and transcription sequencing)则是在scM和T-seq基础上发展起来的,不过这种技术需要将单细胞分离出来,进行处理,检测全基因组染色质可接近性。不同基因组位置的可接近性,或者保护性会影响基因表达,使用scNMT-seq可以发现小鼠胚胎干细胞中表观基因组和转录组之间的新关联。
scMT-seq(another method of simultaneously sequencing single cells’ methylomes and transcriptomes)是同时测序单细胞的甲基化组和转录组的另一种方法,这种是加州大学范国平教授和同济大学薛志刚教授研发的,他们利用这种方法对感觉神经元进行研究,揭示了细胞间的转录组和甲基化组异质性。不过,这些差异大多不是启动子甲基化造成的。举例来说,启动子带CpG岛的基因,表达水平与基因体甲基化正相关。这项研究表明,scMT-seq可以用来检测单细胞中的转录组、甲基化组和单核苷酸多态性信息,进而解析表观遗传学基因调控机制。(同济大学发布单细胞测序新技术)
scTrio-seq(single-cell triple omics sequencing是一种单细胞三重组学测序方法,由北京大学汤富酬教授等人研发,这种全新的单细胞三重组学测序方法在国际上首次从同一个单细胞中实现对三种组学高通量测序信息的同时获取,并从单细胞水平发现肝癌细胞在三种组学上存在密切相互关联的高度异质性。
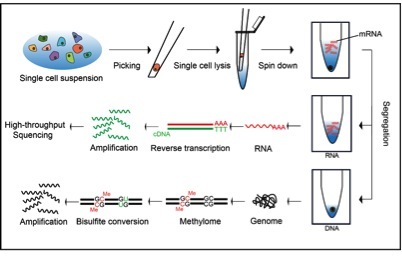
scTrio-seq选择性裂解细胞膜,将细胞质中的mRNA与完整细胞核中的基因组DNA分开。对于scMT-seq,实验人员是利用微量移液管收集细胞核,而在scTrio-seq中,则是通过离心分离细胞核。两种情况中,基因组DNA都是进行的改良亚硫酸盐处理和测序方法来检测甲基化组,而来自细胞裂解物的mRNA则是平行扩增和测序。
“基本上在基因组和转录组数据之间没有交叉污染,”汤教授表示。scTrio-seq使用甲基化组序列数据计算评估基因组拷贝数变异,这种技术已用于分析人结直肠癌样本中的异质性。
蛋白质组和转录组
还有几种技术可以同时测定来自单个细胞的转录物和蛋白质。这些方法为科学家提供了转录后的研究,分析导致蛋白质和转录水平之间的差异。
PLAYR(proximity ligation assay for RNA)技术中,蛋白质被不同金属同位素的抗体标记。同时,RNA转录物被同位素标记的探针结合。利用一种称为质谱流式细胞技术(mass cytometry)的方法测量同位素,并且这种技术也可以同时检测每秒数千个单细胞中的40多种不同的mRNA和蛋白质。
CITE-seq(cellular indexing of transcriptomes and epitopes by sequencing)采用寡核苷酸标记的抗体靶向细胞表面蛋白。这种技术分离并裂解单个细胞,并将它们的mRNA和寡标记抗体与涂有短寡核苷酸序列的磁珠结合。扩增RNA和抗体标签并按大小分离,通过测序定量分析蛋白质和转录物。CITE-seq还可同时检测约100种蛋白质以及数万种RNA转录物。
Marlon Stoeckius是纽约基因组中心的分子生物学家,他是这项技术的研发者。目前Stoeckius正致力于将该方法扩展到细胞内蛋白质,不过这需要固定和透化细胞,这可能会降低RNA质量或导致其从细胞中渗出。
REAP-seq(RNA expression and protein sequencing assay)类似于CITE-seq,采用寡核苷酸交联抗体来检测细胞蛋白质和转录物水平(基于测序)。
REAP-seq和CITE-seq都可以检测很多的转录本,但与PLAYR相比,每次检测的细胞数量更少。
“我认为它们是互补的方法,”PLAYR的首席开发人员Gherardini说,“如果你有一个大的队列或临床研究,像PLAYR这样的东西会更加经济有效”。
CITE-seq还有一个优点就是蛋白质定量可以在通常单细胞RNA测序制备中丢弃的部分进行,因此“对RNA测序文库的质量没有任何损害,”纽约基因组中心的Peter Smibert说,“我们认为如果采用RNA-seq作为读数,那么就可以使用CITE-seq。”


{replyUser1} 回复 {replyUser2}:{content}