4个回答
|
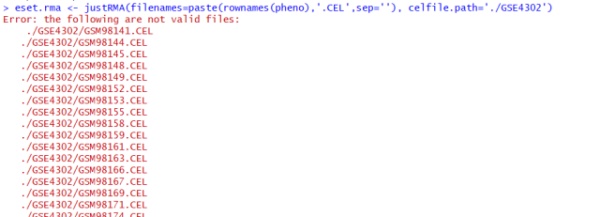
你注意到是部分文件无效了吗?比如,142/143并没有报错。我建议还是要考虑下载的CEL文件不完整。另外,这个GEO有118例样本,比较多,就不建议从原始文件(cel)开始分析,不如从matrix开始,直接做差异表达分析。 |
|
1 .cel文件 我们一般是整个数据集全部预处理然后再提取需要的样本的表达谱,前面预处理过程只用你选的样本与用全部的样本进行标准化是有区别的, 2 我们一般是把.cel文件单独放在一个文件夹,然后justRMA(),没有设置任何参数,都是默认的,如:setwd("路径")#此路径只放整个数据集合的.cel文件,可以是没有解压缩的.cel.gz文件,eset_rma <- justRMA() |
|
看了你的代码和运行报错提示,可以估计存在以下问题:你的cel文件名称部分或全部与实验设计数据框里面不一致。建议GSE4302文件夹里面的cel文件名统一改成GSMXXX.CEL,然后检查必须与实验设计数据框名称一致(这个地方很容易出错),否则RMA无法读取数据的。 |
你的回答
| 提问时间: | 2024-05-02 |
| 浏览量: | 3189 |
| 最近回答: | 2024-05-02 |
相关问题
{title}
投票 {vote}