再创佳绩!上海生科院研究组在CRISPR等领域取得重大进展
碱基编辑器(BE)能够产生靶向的单核苷酸突变,但目前使用的基于APOBEC1的大鼠BE在高度甲基化区域或GpC环境中编辑胞嘧啶的效率相对较低。
2018年8月20日,上海生科院杨力研究组,上海科技大学陈佳研究组及黄行许研究组合作在Nature Biotechnology在线发表题为”Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion“的研究论文,该论文通过筛选各种APOBEC和AID脱氨酶,显示人类APOBEC3A缀合的BEs和设计的具有较窄编辑窗口的版本可以在具有高甲基化水平和GpC二核苷酸含量的区域中介导有效的C-to-T碱基编辑。
已经证明hA3A-BE3及其工程化形式可以在所有检查的环境中全面诱导有效的碱基编辑,包括甲基化DNA区域和GpC二核苷酸。 在未来,hA3A还可以与其他Cas蛋白结合,进一步扩大碱基编辑的范围。
另外,在2018年7月26日,中国科学院上海生科院陈玲玲及杨力合作在Mol Cell在线发表题为“The Biogenesis, Functions, and Challenges of Circular RNAs”的综述,该综述回顾了circRNA生物发生和功能的最新进展,并讨论了circRNA研究中的技术障碍;上海科技大学陈佳研究组及黄行许研究组及上海生科院杨力研究组在Nature Biotechnology发表题为“Base editing with a Cpf1–cytidine deaminase fusion”研究论文,该论文开发了一系列基于CRISPR-Cpf1的碱基编辑工具,它们能够以非常低的indel形式和非C-to-T替换进行有针对性的碱基编辑,并且可以在富含A / T的区域进行编辑;
2018年1月5日,中国科学院-马普计算生物学研究所杨力研究组和中国科学院生物化学与细胞生物学研究所陈玲玲研究组在国际著名学术期刊Molecular Cell发表了题为“N6-methyladenosines modulate A-to-I RNA editing”的最新研究成果,揭示了两种最为普遍存在的RNA水平修饰-腺苷N6位置上的甲基化(m6A)和腺苷至次黄苷碱基编辑(A-to-I editing)-之间的互作关系,阐明了m6A修饰对A-to-I编辑的负向调控作用及其机制。
1.使用人APOBEC3A-Cas9在甲基化区域中进行有效的碱基编辑

将胞苷脱氨酶与Cas9或Cpf1结合的BE已成功应用于进行靶向碱基编辑,包括C-to-T编辑。 据报道,许多人类疾病都是由基因组DNA中的点突变驱动的。 随着最近开发的BEs,这些与疾病相关的点突变可能会得到纠正,从而提供新的治疗选择。 通过分析BEs中理论上可以恢复为胸腺嘧啶的疾病相关T-to-C突变,发现其中~43%在CpG二核苷酸的背景下处于胞嘧啶上。
众所周知,CpG的胞嘧啶通常在哺乳动物细胞中被甲基化,并且胞嘧啶的甲基化强烈抑制由一些APOBEC和AID脱氨酶催化的胞苷脱氨作用。 在这里,上海生科院杨力研究组,上海科技大学陈佳研究组及黄行许研究组合作显示CpG二核苷酸甲基化阻碍了当前BE的C-to-T碱基编辑,同时研究人员并开发了BE在高度甲基化区域中进行有效的C-to-T碱基编辑。
该研究通过筛选各种APOBEC和AID脱氨酶,显示人类APOBEC3A缀合的BEs和设计的具有较窄编辑窗口的版本可以在具有高甲基化水平和GpC二核苷酸含量的区域中介导有效的C-to-T碱基编辑。已经证明hA3A-BE3及其工程化形式可以在所有检查的环境中全面诱导有效的碱基编辑,包括甲基化DNA区域和GpC二核苷酸。 在未来,hA3A还可以与其他Cas蛋白结合,进一步扩大碱基编辑的范围。
原文链接:Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion
2.上海生科院陈玲玲与杨力合作,发表环状RNA最新综述

据报道,通过不同的机制产生了多种环状RNA。例如,它们被鉴定为植物类病毒和肝炎病毒中的环状RNA基因组。家蚕非编码RNA,包括小核仁RNA(snoRNA)和RNase P RNA等。环状RNA中间体也可以在rRNA加工过程中产生。从内含子套索脱支的RNA中间体也可形成环状RNA。尽管存在这些不同形式的环状RNA,但大多数目前研究的环状RNA(circRNA)是由外显子mRNA(前mRNA)反向剪接产生的,其中下游5'剪接位点(ss)与上游3'ss连接。所得到的RNA环在连接位点通过3'-5'磷酸二酯键连接。
circRNAs是在25年前发现的。只有circRNAs极少数是在那个时候发现的,它们往往被认为几乎没有功能性的潜在异常拼接副产品。因为它们的非线性构象和缺乏聚腺苷酸化的(聚(A))尾,circRNAs很少见于下一代RNA测序(RNA-SEQ)分析,其通常富集多聚(A)+ RNA的。仅用非聚(A)RNA进行分析或用RNase R富集环状RNA,RNase R是一种优先消化线性RNA的酶,已经揭示了来自前mRNA回复的circRNA的广泛表达。例如,超过10000 circRNAs已在后生动物中发现,包括蠕,和果蝇,小鼠,猴和人,以及广泛的表达在植物,真菌和原生生物中也发现了circRN。
最近对circRNA生物发生的研究表明,反向剪接由规范的剪接体机制催化,并由内含子互补序列(ICS)和RNA结合蛋白(RBP)调节。新兴研究表明,一些circRNA与神经元功能,先天免疫反应,细胞增殖和多能性有关。在分子水平上,它们通过定量microRNA,隔离蛋白质,调节RNA聚合酶II(Pol II)转录和干扰前mRNA加工参与基因表达。此外,一些内源性circRNA是可翻译的,而另一些可以作为假基因衍生的来源。尽管取得了这些令人鼓舞的进展,但值得注意的是,圆形构象和几乎完整的序列与它们的线性mRNA对应物重叠,这使得对circRNA表达和功能的精确评估具有挑战性。
在这篇综述中,杨力及陈玲玲调查了有关circRNA生物发生和功能调节的最新进展。杨力及陈玲玲还讨论了实验设计及其在circRNA研究中的挑战。
原文链接:The Biogenesis, Functions, and Challenges of Circular RNAs
03.上海科技大学陈佳研究组及黄行许研究组及上海生科院杨力研究组合作开发了一系列基于CRISPR-Cpf1的碱基编辑工具,它们能够以非常低的indel形式和非C-to-T替换进行有针对性的碱基编辑,并且可以在富含A / T的区域进行编辑

各种物种中,通过将 apolipoprotein B mRNA编辑酶,催化多肽样(APOBEC)或激活诱导的脱氨酶(AID)胞苷脱氨酶家族成员与CRISPR-Cas系统组合开发的碱基编辑已经用于靶向C对T碱基编辑【1-5】。然而,使用Cas9切口酶(nCas9)作为目前最活跃的BE中脱氨酶融合伴侣增加了不需要的插入和缺失(插入),非C-T碱基置换的频率,限制编辑富含G / C的PAM序列的区域【6,7】。
Cpf1(Cas12a)是另一种Cas蛋白,它与Cas9的不同之处在于:Cpf1需要富含T的PAM序列(TTTV)用于目标DNA的识别【8,9】; Cpf1(CRISPR RNA(crRNA))的sgRNA比Cas9短;并且Cpf1切割位点相对于间隔区DNA中的PAM序列位于远端和下游,而不是与Cas9的近端和上游【10,11】。与Cas9相比,Cpf1也诱导更少的脱靶(OT)切割基因组【12,13,14】。
为了解决以上问题,上海科技大学陈佳研究组及黄行许研究组及上海生科院杨力研究组合作通过将大鼠胞嘧啶脱氨酶APOBEC1融合到无催化活性的Lachnospiraceae细菌Cpf1上,开发了基于CRISPR-Cpf1的碱基编辑工具。 碱基编辑器可识别富含T的PAM序列,并催化人类细胞中的C至T转换,同时诱导低水平的插入缺失,非C至T替换和脱靶编辑。
该研究首次开发了一系列基于CRISPR-Cpf1的碱基编辑工具,它们能够以非常低的indel形式和非C-to-T替换进行有针对性的基础编辑,并且可以在富含A / T的区域进行编辑。 未来,预计其他Cpf1酶(例如,识别TTN PAM8的FnCpf1)或工程化的Cpf1 可用于进一步增强dCpf1的碱基编辑作用。
原文链接:Base editing with a Cpf1–cytidine deaminase fusion
参考文献:
1.Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A. & Liu, D.R. Nature 533, 420–424 (2016).
2. Nishida, K. et al. Science 353, aaf8729 (2016).
3. Kim, K. et al. Nat. Biotechnol. 35, 435–437 (2017).
4. Zong, Y. et al. Nat. Biotechnol. 35, 438–440 (2017).
5. Li, G. et al. Protein Cell 8, 776–779 (2017).
6. Kim, Y.B. et al. Nat. Biotechnol. 35, 371–376 (2017).
7. Kleinstiver, B.P. et al. Nature 523, 481–485 (2015).
8. Zetsche, B. et al. Cell 163, 759–771 (2015).
9. Yamano, T. et al. Cell 165, 949–962 (2016).
10. Stella, S., Alcón, P. & Montoya, G. Nature 546, 559–563 (2017).
11. Swarts, D.C., van der Oost, J. & Jinek, M. Mol. Cell 66, 221–233.e224 (2017).
12. Kim, D. et al. Nat. Biotechnol. 34, 863–868 (2016).
13. Kleinstiver, B.P. et al. Nat. Biotechnol. 34, 869–874 (2016).
14. Yan, W.X. et al. Nat. Commun. 8, 15058 (2017).
04.上海生科院杨力研究组合作发表不同RNA修饰间的互作调控

迄今为止,多达100多种的RNA修饰已在体内被发现,其中m6A修饰及A-to-I编辑是存在于(m)RNA上最普遍的两种RNA水平的表观修饰,且都发生于腺苷(adenosine, A)上。这两种A上的RNA表观修饰在催化原理和发生位置上存在着很大的不同:m6A主要由甲基化酶复合体(METTL3和METTL14)催化发生,并由去甲基化酶(FTO和ALKBH5)可逆调节去甲基,其主要发生在单链RNA的A碱基上;而A-to-I编辑主要由ADAR酶介导,其主要发生在位于双链RNA的A碱基上,尚没有可逆的编辑酶被发现。因此,基于它们在催化原理和发生位置上的差异,推断m6A和A-to-I这两种最为普遍的RNA表观修饰一般来讲不会竞争在同一个A碱基位置发生。但是,它们之间是否存在其它的互作关系?

部分实验验证
在这项最新的工作中,研究人员利用计算和实验相结合的系统研究方法,对m6A阳性和m6A阴性的RNA-seq数据进行A-to-I编辑的比较分析,首先揭示了m6A修饰与A-to-I编辑之间存在着一定的负相关关系;通过分析m6A甲基化酶METTL3和/或METTL14敲除样本中的A-to-I编辑进一步揭示了m6A修饰对A-to-I编辑的负向调控作用;最后,通过对多个内源转录本和构建的报告质粒在正常条件和METTL3/METTL14双敲条件下进行比较分析,发现ADAR1与m6A阳性转录本的结合能力较弱,而在METTL3/METTL14双敲抑制m6A时,ADAR1与m6A阴性转录本的结合能力则显著提高,这提示m6A修饰对A-to-I编辑的负向调控作用可能是通过调节其与ADAR1的结合能力而实现的。
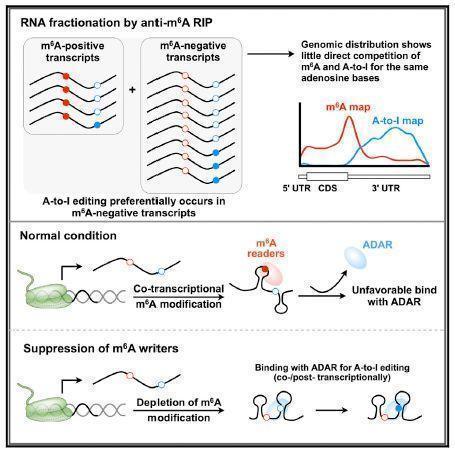
作用模型
该项研究在杨力研究员和陈玲玲研究员的共同指导下,由计算生物学所博士后向剑锋、博士研究生杨钦和生化与细胞所博士研究生刘楚霄共同完成,得到了国家基金委、科技部、中科院以及HHMI基金会的经费支持。杨力研究员长期从事计算生物学和生物大数据研究,与陈玲玲研究员合作揭示了A-to-I编辑在不同转录组中的动态变化调控(Zhu et al, MBC Genomics 2013)以及A-to-I编辑酶ADAR在miRNA成熟过程中的全新作用(Chen et al, Cell Res 2015),而这项最新的不同RNA修饰之间的互作研究为全面揭示复杂RNA表观修饰调控提供了新的思路和基础。
来源:iNature
版权及免责声明:本网站所有文章除标明原创外,均来自网络。登载本文的目的为传播行业信息,内容仅供参考,如有侵权请联系答魔删除。文章版权归原作者及原出处所有。本网拥有对此声明的最终解释权。


{replyUser1} 回复 {replyUser2}:{content}